富集组蛋白或转录因子(TF)的复合DNA在体内,然后下一代测序提供了一个有利的工具,研究全基因组蛋白质-DNA相互作用。它允许分析与活细胞中DNA序列结合的特定蛋白质。这样的分析要求该方法可靠地鉴定真正的靶蛋白富集区域,特别是来自有限的细胞样品。这些样品可以包括从组织中分离的稀有细胞群、从整个细胞群分选的特定细胞和原代培养的亚群细胞如胚胎细胞。此外,该方法应确保富集的DNA含有最少的背景,并且蛋白质-DNA结合区的作图具有最小偏差和高分辨率。已经使用了很长一段时间来实现该目标的主要方法是染色质免疫沉淀,然后是测序(ChIP-Seq)。然而,ChIP的主要局限性在于,它需要1)大量的输入材料,细胞或组织,以产生超过背景噪声的足够强的信号;和2)在初始固定步骤期间交联。有几种先进的方法可用于ChIP-Seq,具有减少的细胞数量或增加的分辨率。这些方法包括ChIP-exo和ChIPmentation。ChIP-exo提供了高分辨率的映射,但耗时并且需要充足的输入细胞供应。虽然ChIP片段化使用转座酶和测序相容的衔接子以使得能够在ChIP过程中整合连接,但其遵循传统上缓慢(2天)的ChIP程序并且不能实现高分辨率作图。CUT RUN(Cleavage Under Target Release Using Nuclease)是为了将捕获的靶蛋白/DNA复合物从有限的生物材料中释放出来,用于蛋白质-DNA相互作用的作图,它显著提高了作图分辨率。然而,CUT RUN需要昂贵的PA/Mnase融合蛋白,其具有显著的A/T序列偏差,导致靶蛋白相互作用的DNA区域谱严重受MNase消化水平的影响。
据今年3月份发布在ResearchSquare的一项研究,佛罗里达的科学家使用CUT&RUN技术,建立了眼球晶状体中HIF1α结合位点的全基因组图,文章链接:https://assets.researchsquare.com/files/rs-247248/v1/83c34fd6-e42a-491b-8828-247ace7a4fb8.pdf?c=1631877386

我们知道,基因表达可以通过许多不同的机制进行调节,包括转录因子、组蛋白和其他DNA结合蛋白。绘制出DNA和蛋白质之间的相互作用图谱,对于理解基因激活或抑制以及其他细胞过程至关重要。
传统上,可以通过进行染色质免疫沉淀和测序(ChIP-seq)来分析这些相互作用。但是这种方法需要大量的起始材料才能产生足够强的信号以消除背景噪声,并且经常返回假阳性信号。同时,ChIP-seq还需要优化的超声处理来破碎染色质,这可能非常耗时并可能导致样品损失。
在最近的一项研究中,佛罗里达大西洋大学和国家糖尿病、消化和肾脏疾病研究所的科学家们利用CUT&RUN技术建立了一个功能性全基因组图谱,该图谱是一种称为缺氧诱导因子的蛋白质复合物的特异性结合位点—HIF1α,位于人眼的晶状体中。该团队表示选择CUT&RUN而不是ChIP-seq分析,是因为前者不需要DNA-蛋白质交联。此外,与ChIP-seq相比,前者成功实验所需的细胞计数极低。
正常而言,随着出生后眼睛的发育,提供健康血液供应的血管随着时间的推移开始萎缩。结果,眼睛中的氧气含量稳步下降,造成缺氧环境。HIF1α作为一种蛋白质复合物,可调节身体对缺氧环境的反应,这意味着它可以在晶状体细胞发育和体内平衡中发挥作用。
研究人员首先选择培养10天左右的大的WhiteLeghorn鸡胚胎中分离出晶状体细胞。收获的细胞用称为DMOG的HIF1α激活剂处理4小时,并用ConA包被的磁珠固定细胞核。之后,样品被分为两组:一组用HIF1α抗体处理,另一组用IgG抗体处理。将pA/MNase添加到样品中以启动碎裂。这种融合蛋白在与感兴趣的蛋白质相互作用的特定区域剪切DNA——在这种情况下,该蛋白质是HIF1α。通过向样品中加入CaCL2溶液来激活MNase,将DNA片段释放到上清液中。CUT&RUN片段用蛋白酶K处理1小时,然后分离DNA进行测序。
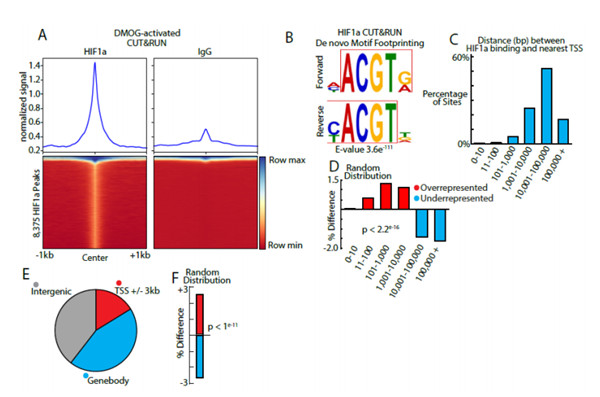
在HIF1α激活后,CUT&RUN在原代晶状体细胞中鉴定了8000多种HIF1α-DNA特异性复合物。他们能够使用ATAC-seq和RNA-seq验证并完成结合位点的映射,这表明这些复合物中约有1200个紧密聚集在染色质可及区域。进一步的分析揭示了526个基因的激活或抑制,其中116个基因在转录起始位点10kB内显示HIF1α结合位点。
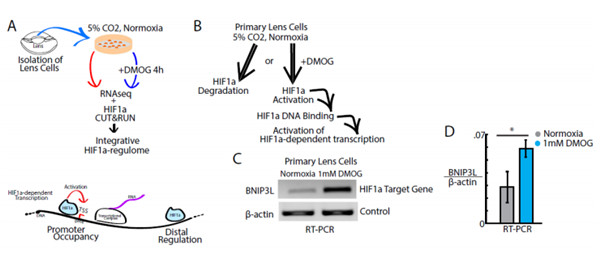
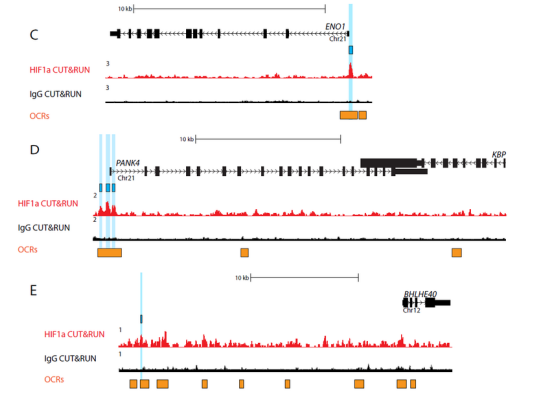
该实验中收集的数据有助于建立眼晶状体中HIF1αDNA复合物的第一个功能图,同时也强调了健康晶状体发育对HIF1α的需求。最终,CUT&RUN技术的这种成功应用,有助于接下来的大量研究,重点是了解某些蛋白质可能对基因表达的作用。
产品推荐
艾美捷科技作为表观遗传领域专业的解决方案供应商,为您推荐EpiGentek品牌的EpiNext EpiNext CUT&RUN快速套件,货号P-2028,该试剂盒以优惠价格从低输入提供无超声破碎,以可靠地识别真正的目标蛋白质富集区域并实现高分辨率定位。可满足您快速富集蛋白质结合的DNA并绘制全基因组蛋白质/DNA相互作用的图谱的需求。
从哺乳动物细胞或分离的细胞核/染色质开始进行成功的CUT RUN所需的一整套优化试剂。该试剂盒被设计成快速富集具有来自低输入细胞/染色质的蛋白质(组蛋白或转录因子)的特定DNA复合物,并通过下一代测序使用Illumina平台或其他方法如qPCR鉴定或绘制体内蛋白质-DNA相互作用。创新的工作原理、优化的方案和试剂盒的组件允许捕获靶向蛋白质/DNA复合物与最小化的非特异性背景水平。
| 货号 | 名称 | 厂家 | 时长 |
| P-2028 | EpiNext CUT&RUN快速套件 EpiNext CUT&RUN Fast Kit | Epigentek | <3小时 |
结果展示
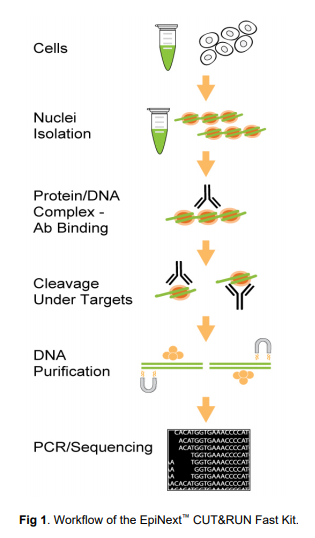
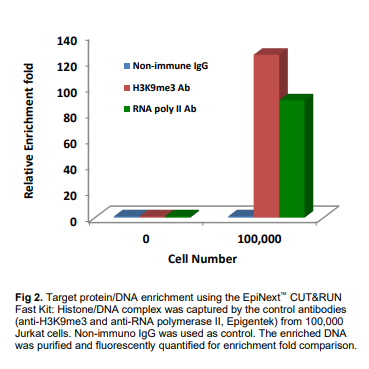
EpiNext CUT&RUN快速套件优势:
1、高富集:使用独特的核酸切割酶混合物,其具有低序列偏好,以同时片段化染色质并切割/去除靶蛋白/DNA复合物两端的任何DNA序列,而不影响靶蛋白占据的DNA。富集的DNA 20 bp可被高效、快速地回收。因此,靶蛋白富集区域可以可靠地获得和在高分辨率作图中鉴定。
2、低输入材料:稳健的无超声破碎、未结合的DNA切割和免疫捕获都在相同的单管中进行处理,具有珠上连接。该方法使用细胞和组织两者,并且允许靶蛋白的最大降解保护,且样品损失最小。因此,输入细胞量可以少至500个细胞,或者染色质量可以低至0.1 ?g。
3、最小背景:原位切割靶蛋白质/DNA复合物的两(2)末端中未结合的DNA序列使得免疫捕获背景最小化,从而允许具有1000万个读段的测序数据分析<。
4、快速、简化的程序:从细胞到文库DNA的过程少于3小时。
5、非常方便:该套件包含切割运行每个步骤所需的所有组件&,从而使EpiNext?切割&运行快速套件最方便,结果可靠且一致。

微信扫码在线客服