【前情回顾】Epigentek : 染色质研究
【前情回顾】Epigentek染色质可及性分析试剂盒,解码基因组的创新工具
表观遗传修饰在基因调控中发挥着关键作用,对多种生物过程有着深远的影响。染色质免疫沉淀(ChIP)一直是研究细胞中表观遗传修饰和蛋白质-DNA相互作用的金标准。然而,传统的ChIP存在一些局限性,包括需要大量的起始材料、处理时间长以及分辨率较低。两种新兴技术——靶向切割与核酸酶释放(CUT&RUN)和靶向切割与标记(CUT&Tag)——作为ChIP的有力替代方法脱颖而出。更重要的是,最近,表观遗传研究专家EpigenTek公司进一步改进了CUT&RUN-Fast方法,推出了新的CUT&LUNCH(Cleavage Under Target and Liberate Unique Nucleic Complex Homogeneously,靶向切割与均匀释放独特核酸复合物)技术,该技术具有极其快速的实验流程、进一步提升的特异性以及最小化的背景信号。本文将比较传统的ChIP与CUT&RUN和CUT&Tag,以及新兴的CUT&LUNCH技术,并讨论它们的优缺点。
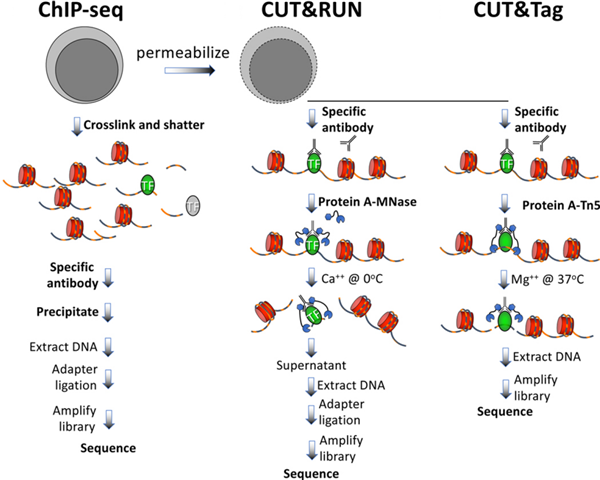
在染色质免疫沉淀(ChIP)过程中,首先将蛋白质与DNA进行交联。随后,固定后的染色质被片段化,接着利用抗体特异性地免疫沉淀目标蛋白质(例如,转录因子、组蛋白修饰、染色质重塑因子)及其相关联的DNA片段。最后,通过纯化和测序这些DNA片段,可以确定目标蛋白质结合的基因组区域。尽管染色质免疫沉淀(ChIP)是研究蛋白质-DNA相互作用的有力工具,但它也存在一些缺点:
(1) 大多数ChIP实验需要大量的起始材料,这在处理有限的细胞数量或稀有细胞群体时可能是一个挑战。
(2) 该技术过程繁琐,涉及多个步骤,包括交联、片段化和免疫沉淀,整个过程可能需要数天才能完成。
(3) ChIP的分辨率有限,因为免疫沉淀的DNA片段可能包含未被目标蛋白质结合的区域。
CUT&RUN和CUT&Tag技术的开发旨在解决这些不足。这两种技术均在完整细胞或细胞核中进行,无需固定处理,通过核酸酶在特异性抗体结合位点切割染色质,并直接捕获蛋白质-DNA复合物。CUT&RUN操作流程包括抗体介导的核膜通透化,随后由融合蛋白在抗体结合的目标蛋白区域进行DNA切割。融合蛋白由切割域(如微囊藻核酸酶或MNase)和结合抗体的蛋白A/G(pAG)域组成。抗体特异性结合目标蛋白,pAG-MNase融合蛋白与抗体结合,并在蛋白质-DNA相互作用位点切割染色质,从而释放蛋白质-DNA复合物。CUT&Tag与CUT&RUN类似,但在切割过程中加入了转座酶,该酶将测序接头插入切割后的DNA片段中。随后对DNA进行扩增和测序。CUT&RUN和CUT&Tag是有效的技术,有望克服ChIP的一些局限性:1.它们所需的起始材料更少。CUT&RUN可以在低至1000个细胞的情况下进行,而CUT&Tag甚至可以在少于100个细胞的情况下进行;2.处理时间更短,CUT&RUN的完成时间不到半天,CUT&Tag的完成时间不到一天;3.CUT&RUN和CUT&Tag的分辨率高于ChIP,因为它们在细胞内原位切割染色质,从而可以直接捕获蛋白质-DNA复合物;4. CUT&Tag是一种单管反应,简化了操作流程,并减少了所需的起始材料;5. 这两种技术的背景噪声均低于ChIP,从而实现了更高的信噪比。
随着CUT&RUN和CUT&Tag的不断优化和完善,它们有可能在表观遗传学领域得到更广泛的应用。特别是,CUT&RUN和CUT&Tag的高分辨率和低背景噪声使它们成为研究稀有细胞群或低丰度蛋白质的有吸引力的选择。此外,CUT&Tag的单管反应可能对高通量应用特别有用,例如单细胞测序。CUT&RUN- fast是改进的CUT&RUN技术,CUT&RUN- fast技术采用了一种新颖独特的核酸切割酶混合物,具有低序列偏倚,可以同时切割染色质并切割/去除靶蛋白/DNA复合物两端的任何DNA序列,而不会影响靶蛋白所占据的DNA,从而提高了特异性和分辨率。
货号 | 名称 | 应用 |
EpiNext CUT&RUN-Fast Kit | 用于制备蛋白质-DNA相互作用分析样品,具有比ChIP更好的信噪比。在这个反应中,细胞核从细胞中分离出来。目标蛋白- dna复合物与感兴趣的chip级抗体结合/捕获。利用一种独特的核酸裂解酶混合物,染色质被切碎,靶蛋白/DNA复合体两端的DNA序列被切割/去除。同时,目标蛋白所占据的DNA序列不受影响。然后纯化和洗脱目标蛋白质结合的DNA。富集的DNA可用于各种方法分析蛋白质-DNA相互作用,特别是下一代测序。 | |
P-2032 | EpiNext cTIP (CUT&Tag-In-Place)-Sequencing Kit | 用于高分辨率染色质分析与所有一体化的蛋白质-dna复合物富集和NGS文库生成。在这个反应中,细胞核从细胞中分离出来。目标蛋白- dna复合物与感兴趣的chip级抗体结合/捕获。利用一种独特的核酸裂解酶混合物,染色质被切碎,靶蛋白/DNA复合体两端的DNA序列被切割/去除。在此期间,目标蛋白所占据的DNA序列不受影响。接头连接到珠上的靶蛋白结合DNA片段上。然后释放连接的DNA,纯化,并用高保真PCR混合扩增用于文库DNA构建。然后DNA被清洗、释放和洗脱。 |
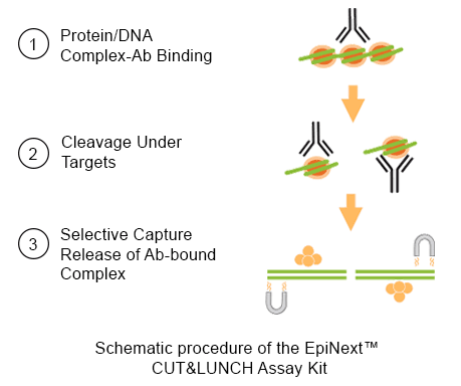
近来,EpigenTek进一步完善了CUT&RUN-Fast方法,采用新的靶下切割和均匀释放独特的核酸复合物(CUT&LUNCH),该方法具有极快的操作流程,进一步增强了特异性,并最小化了背景。
货号 | 名称 | 应用 |
EpiNext CUT&LUNCH Assay Kit | 用于目标蛋白- dna复合物的超快速、高特异性富集。在这个实验中,细胞被渗透并暴露于感兴趣的chip级抗体。使用一种独特的核酸切割酶混合物,在目标染色质区域的两端的DNA序列被切割/去除。释放的非特异性蛋白质- dna复合物被消除,只有抗体结合的复合物将被选择性地恢复。捕获的蛋白质/DNA复合物中的DNA片段被纯化,可直接用于基因特异性qPCR或DNA文库构建,以分析蛋白质与DNA之间的相互作用。 | |
EpiNext CUT&LUNCH-Seq Kit | 用于超快速,高特异性富集目标蛋白- dna复合物与文库准备一体化。在这个实验中,细胞被渗透并暴露于感兴趣的chip级抗体。使用一种独特的核酸切割酶混合物,在目标染色质区域的两端的DNA序列被切割/去除。释放的非特异性蛋白质- dna复合物被消除,只有抗体结合的复合物将被选择性地恢复。接头连接到从捕获的蛋白质/DNA复合物中纯化的目标DNA片段。然后用高保真PCR组合扩增连接的DNA,用于构建文库DNA,利用NGS分析蛋白质和DNA之间的相互作用。 |

微信扫码在线客服